Книга: Самое грандиозное шоу на Земле
Молекулярное сопоставление
| <<< Назад Что сделал бы Дарси Томпсон, будь у него компьютер |
Вперед >>> Молекулярные «часы» |
Молекулярное сопоставление
Дарвин, конечно, не мог знать, что сравнительные доказательства станут еще более убедительными, если дополнить доступные ему анатомические сравнения молекулярной генетикой.
Так же, как скелет позвоночных остается неизменным при изменении отдельных костей, а экзоскелет всех ракообразных одинаков независимо от формы составляющих его «трубок», код ДНК одинаков у всех живых существ — меняются только отдельные гены. Это поразительный факт, яснее всех других демонстрирующий, что все живые существа происходят от общего предка. Не только генетический код, но, как мы видели в главе 8, вся генно-белковая система, поддерживающая жизнь, одинакова у всех животных, растений, грибов, бактерий, архей и вирусов. Меняется лишь закодированная информация, сам код остается неизменным. И если посмотреть на то, что закодировано, на последовательности генов разных существ, мы снова увидим генеалогическое древо, которое получалось при сравнении скелета позвоночных, скелетов ракообразных, да даже необязательно скелета — годятся все морфологические признаки.
Допустим, мы захотели узнать, насколько близки друг другу два вида, например, ежи и обезьяны. Идеальное решение проблемы — взять полные генетические коды этих видов и сравнить их построчно и побуквенно, как библеист сравнивал бы два рукописных варианта Книги пророка Исайи. Но это дорого и отнимет много времени. Работа над проектом «Геном человека» шла десять лет, сотни человеко-лет труда. Хотя сейчас это можно сделать намного быстрее, проект «Геном ежа» все равно будет недешевым и не слишком быстро осуществимым.
«Геном человека» — один из проектов, которые, подобно высадке на Луне или постройке Большого адронного коллайдера, вызывают у меня чувство гордости за человечество. Я рад и тому, что недавно закончены секвенирование генома шимпанзе и аналогичные проекты для других видов. Если мы и дальше будем двигаться вперед такими темпами (см. обсуждение закона Ходжкина ниже), довольно скоро сравнительный анализ и оценка родства на основе полной генетической информации станут экономически доступными. Пока же наши возможности ограничены сравнительным анализом частей генома, но и этот метод дает отличные результаты.
Для сравнения достаточно выбрать у двух видов несколько генов (или белков, строение которых известно непосредственно из генетических последовательностей) и сравнить их. Мы вскоре к этому вернемся. Однако давно известны и другие, более грубые, методы автоматизированного сравнения, которые на удивление хорошо работают. Один из них основан на использовании иммунной системы кроликов (подойдет любое другое животное, но кролики отлично справляются). Защищаясь от патогенов, иммунная система кроликов вырабатывает антитела к любому чужеродному белку[153], обнаруженному ею в крови. Как анализ антител в моей крови свидетельствует о том, что недавно меня мучил кашель, точно так же иммунный ответ кролика говорит о том, что происходило с ним в прошлом. Антитела в крови кролика — своего рода история случавшихся с ним неприятностей, включая искусственное введение белков. Если, например, впрыснуть в кровь кролика белок от шимпанзе, иммунная система выработает антитела для атаки этого белка на случай, если он снова появится в крови. Но предположим, что в следующий раз мы введем близкий, но не идентичный белок, например от гориллы. Опыт встречи с белками шимпанзе частично защитит кролика от белков гориллы, но реакция будет менее выраженной. Поможет он и от белка кенгуру, но защитная реакция будет еще слабее, поскольку кенгуру значительно дальше от шимпанзе, чем горилла. Следовательно, сила иммунного ответа может служить мерой родства нового белка с исходным, сформировавшим ответ. Именно так Винсент Сэрич и Алан Уилсон из Калифорнийского университета в Беркли показали в 1960-х, что человек и шимпанзе гораздо ближе, чем считалось.
Есть и методы, использующие для сравнения видов гены, а не кодируемые ими белки. Один из самых старых и эффективных прямых методов — так называемая гибридизация ДНК. Именно этот метод стоит за широко известными утверждениями типа «у человека и шимпанзе более 98 % общих генов». Кстати, конкретный смысл таких цифр нуждается в уточнении: 98 % чего именно у нас общего с шимпанзе? Точный ответ зависит от размера единиц счета. Попробую пояснить это с помощью простой аналогии. Она интересна тем, что отличие от нее действительности говорит не меньше, чем сходство.
Допустим, у нас есть два варианта одной книги, и мы хотим сравнить их. Например, это каноническая Книга пророка Даниила и свиток, недавно обнаруженный в какой-нибудь пещере на берегу Мертвого моря. Каков процент одинаковых глав в двух текстах? Скорее всего, нулевой: отличия одной буквы достаточно, чтобы различались главы. А каков процент совпадающих предложений? Вероятно, заметно выше. Процент совпадающих слов еще выше, поскольку в слове букв меньше, чем в предложении. Однако слова, отличающиеся одной буквой — все-таки разные слова. Если мы сравним два текста, буква за буквой, то процент совпадений будет еще выше, чем со словами. Итак, оценка ничего не значит, пока мы не определим, что именно мы сравниваем: главы, параграфы, предложения, слова или буквы. Точно так дело обстоит со сравнением ДНК двух видов: если сравнивать целые хромосомы, совпадений не будет, поскольку любого отличия достаточно, чтобы хромосомы отличались.
Часто упоминаемые 98 % общего генетического материала людей и шимпанзе — это не хромосомы или целые гены, а совпадающие «буквы ДНК» (пары оснований). Однако все не так просто. Если сравнивать буквы в текстах по порядку, то вставленная или пропущенная, а не просто несовпадающая буква сдвигает все стоящие после нее буквы. Ясно, что такого рода несоответствие нас не интересует и должно быть предусмотрено. Глаз исследователя, сравнивающего варианты Книги пророка Даниила, исключает такие случаи автоматически (кстати, алгоритм такого исключения нетривиален). Как добиться того же в отношении ДНК? Настал момент оставить нашу аналогию и вернуться к ДНК, поскольку это тот случай, когда аналогия оказывается сложнее действительности.
Если ДНК постепенно нагревать, в некоторый момент (около 85 °C) связи между двумя цепочками двойной спирали разорвутся и цепочки разойдутся. Данную температуру можно считать своего рода точкой плавления. Если теперь дать цепочкам ДНК остыть, они (или их фрагменты) начнут спонтанно образовывать двойные спирали по обычным правилам связывания пар оснований. Можно было бы предположить, что повторное связывание будет происходить между теми же, полностью совместимыми, фрагментами. Так и вправду могло быть, но в действительности фрагменты ДНК найдут себе не обязательно те же пары, что прежде. Более того, если добавить фрагменты ДНК другого вида, фрагменты цепочек разных видов могут объединяться точно так же, как цепочки спирали одного вида. А почему бы и нет? Это удивительное, но очевидное следствие революционного постулата молекулярной биологии Уотсона-Крика: ДНК — это просто ДНК. Ей нет дела, чья она: человека, шимпанзе или яблока. Фрагменты с удовольствием объединятся с любым комплементарным фрагментом, который найдут. Тем не менее сила связи непостоянна. Одиночные фрагменты цепочки ДНК связываются с другими фрагментами тем сильнее, чем более они подходят друг другу, и наоборот. Причина в том, что чем менее подходят друг другу «буквы» фрагментов (основания Уотсона-Крика), тем больше несоответствий, тем слабее связь, как у застежки-«молнии» с отсутствующими зубцами. Как измерить силу связи после того, как фрагменты цепочек разных видов найдут подходящие пары и свяжутся? Мы нагреем их до точки плавления! Точка плавления обычной двойной спирали с полностью соответствующими цепочками, например, ДНК человека — около 85 °C. Но когда связь слабее, например, когда цепочка ДНК человека связана с цепочкой ДНК шимпанзе, для разделения оказывается достаточно меньшей температуры. А для цепочек видов, дальше отстоящих друг от друга, например человека и рыбы либо жабы, «температура плавления» еще ниже. Разность «температур плавления» спирали, составленной из цепочек одного вида и смеси с цепочками другого, будет нашей мерой генетической близости этих видов. Правило звучит примерно так: снижение температуры на 1 °C эквивалентно 1 % несовпадающих «букв» ДНК (или отсутствию 1 % зубцов «молнии»).
Конечно, у этого метода есть сложности с нетривиальными решениями, в которые я не вдавался. Например, если смешать ДНК человека и шимпанзе, большинство фрагментов при остывании объединится с фрагментами цепочек своего вида. Как отделить «гибридную» ДНК, «температуру плавления» которой надо измерить, от двух чистых ДНК? Ответ: используя радиоактивные метки (подробный рассказ увел бы нас далеко от темы). Сейчас нам важно только то, что именно гибридизация ДНК позволяет получить оценки генетической близости наподобие 98 % для человека и шимпанзе, которые в полном соответствии с теорией уменьшаются по мере отдаления видов.
Самый современный (и затратный) метод определения генетической близости пар соответствующих генов — прямое считывание и сравнение генов тем же способом, что в проекте «Геном человека». Сравнение геномов целиком по-прежнему стоит очень дорого, но сравнение отдельных наборов генов дает хорошие результаты и все чаще применяется на практике.
Каким бы методом измерения степени близости видов мы ни пользовались, будь то кроличьи антитела, «точки плавления» или прямое сравнение, следующий шаг будет одним и тем же. Получив числа, означающие степень близости видов, мы поместим эти числа в таблицу. Строки и столбцы — названия видов, написанные в одинаковом порядке; на их пересечении, в клетках — степень сходства. Таблица получится треугольной (рассеченный наискосок квадрат), потому что, например, сходство между человеком и собакой точно такое, как между собакой и человеком. И, если заполнить все клетки таблицы, ее половины по обе стороны диагонали окажутся зеркальным отражением друг друга.
Какой результат мы ожидаем увидеть? Эволюционная модель предсказывает, что в клетке «человек/шимпанзе» сходство будет выше, чем в клетке «человек/собака». В клетках «человек/собака» и «шимпанзе/собака» значения должны быть одинаковыми, поскольку степень родства человека с собакой та же, что у шимпанзе с собакой. То же значение должно оказаться в клетках «обезьяна/собака» и «лемур/собака». Причина в том, что человек, шимпанзе, обезьяны и лемуры связаны с собакой через одного общего предка — раннего примата, который, скорее всего, был немного похож на лемура. Так же должны соотноситься клетки «человек/кошка», «шимпанзе/кошка», «обезьяна/кошка» и «лемур/кошка», потому что кошек и собак с приматами связывает общий предок всех хищников. Во всех клетках, связывающих, например, кальмара с млекопитающими, должно стоять примерно одно и то же число, причем маленькое. И неважно, о каком млекопитающем идет речь: они одинаково далеки от кальмара.
Но это теория, а практика иногда сильно от нее отличается. Если мы увидим противоречие, оно будет аргументом против эволюционной теории. Однако в действительности (с учетом статистической погрешности) мы наблюдаем именно то, что предсказывает эволюционная теория. Иными словами, если приписать значения сходства между парами видов к ветвям нашего дерева, то все сойдется. Конечно, абсолютного совпадения нет, но численные оценки в биологии редко даются с точностью выше некоторого приближения.
Доводы сравнительного анализа ДНК или белков позволяют сделать вывод о том, какие пары видов являются близкородственными. Однако сила этого метода как доказательства эволюционной теории заключается в возможности построить дерево генетического родства по каждому гену. Важнейший результат заключается в том, что все такие деревья во многом одинаковы. Именно такой результат и должен был получиться, если перед нами генеалогическое древо. И совсем не то, что мы ожидали, если бы Творец выбирал подходящие белки то там, то здесь, вне зависимости от первоначального носителя белка.
Первое масштабное исследование этого рода было выполнено группой новозеландских генетиков под руководством профессора Дэвида Пенни[154]. Группа Пенни выбрала пять генов, которые пусть не идентичны у всех млекопитающих, но достаточно схожи для того, чтобы у всех называться одинаково. Это гены гемоглобина А, гемоглобина В (он придает крови красный цвет), фибринопептида А, фибринопептида В (фибринопептиды отвечают за свертывание крови) и цитохрома С, играющего важную роль в биохимии клетки. Для сравнения были выбраны одиннадцать млекопитающих: макак-резус, овца, кенгуру, крыса, кролик, собака, свинья, человек, лошадь, корова и шимпанзе.
Пенни с коллегами подошли к задаче статистически. Они решили оценить вероятность того, что две молекулы могут дать одно и то же генеалогическое древо случайно. Поэтому они перебрали все деревья, которые можно построить для выбранных одиннадцати видов. Это число оказалось на удивление большим. Даже если ограничиться бинарными деревьями (в каждом узле ствол делится на две ветви, то есть дает двух потомков), то количество вариантов превысит 34 миллиона. Ученые терпеливо перебрали эти деревья, сравнив каждое с остальными 33999999 вариантами. То есть, конечно, они не делали этого: ушло бы слишком много времени. Вместо этого они придумали изощренный статистический эквивалент этих чудовищных по объему вычислений.
Метод приблизительно такой. Возьмем первые пять генов, например, гемоглобина А (я буду использовать здесь название белка, который кодируют гены) и найдем среди миллионов деревьев самое экономное для гемоглобина А. Под экономным понимается дерево, которое требует минимального числа эволюционных изменений. Например, все деревья, где кенгуру ближе к человеку, чем к шимпанзе, далеки от экономного: они требуют очень большого числа изменений, чтобы у кенгуру и человека нашелся близкий общий предок. Если бы гемоглобин А умел говорить, он рассказал бы примерно следующее:
Это очень неэкономное дерево. Оно требует не только огромного числа мутаций, чтобы получить два таких разных, но в то же время близкородственных организма, как кенгуру и человек, но еще и огромного числа мутаций, чтобы человек и шимпанзе оказались столь похожими с моей, гемоглобиновой, точки зрения. Я против этого варианта.
В отношении каждого из 34 миллионов деревьев гемоглобин А вынес свой вердикт, отобрав в итоге десятка полтора самых экономных деревьев. О каждом из них он отозвался бы приблизительно так:
В этом варианте человек и шимпанзе — близкие родственники, коровы и овцы также, а кенгуру находятся где-то на своей ветке. Это хорошее дерево, потому что мне не придется выполнять почти никакой мутационной работы для того, чтобы объяснить эволюционные изменения. Это очень экономное дерево. Я голосую «за».
Конечно, было бы приятно, если бы гемоглобин А и остальные гены «проголосовали» за одно и то же экономное дерево, но это было бы слишком. Понятно, что среди 34 миллионов деревьев найдется несколько достаточно симпатичных гемоглобину А.
Однако не будем забывать о гемоглобине В, цитохроме С и других белках. Когда все они голосовали за своих кандидатов из 34 миллионов деревьев, можно было бы ожидать, что их вкусы окажутся разными. Могло, например, оказаться, что с точки зрения цитохрома С человек и вправду гораздо ближе к кенгуру, чем к шимпанзе. В отличие от гемоглобина А, цитохром С мог бы, например, сказать, что ему не составит больших (мутационных) затруднений превратить овцу в обезьяну или сделать коров и кроликов близкими родственниками. Если придерживаться креационистской гипотезы, этому ничто не препятствует. Но Пенни и его коллеги обнаружили, что все пять генов голосуют практически одинаково (более того, они оценили вероятность того, что результат мог случайно оказаться таким же). Все гены «проголосовали» за одни и те же деревья, чего следовало ожидать эволюционисту, предполагающему существование одного дерева, связывающего одиннадцать животных в генеалогическое древо — древо эволюционных изменений. Самое любопытное, что дерево, победившее в «голосовании» генов, оказалось именно тем вариантом, который зоологи получили независимо, на основании анатомических и палеонтологических данных.
Результаты исследования Пенни были опубликованы в 1982 году, то есть довольно давно. С тех пор было проделано огромное число детальных исследований на основе конкретных генетических последовательностей разных животных и растений. Консенсусное древо, которое удовлетворяет всех участников «голосования», теперь охватывает куда больше пяти генов и одиннадцати видов оригинального исследования Пенни. Это был просто изящный пример, оказавшийся очень сильным аргументом из-за примененной статистической методологии.
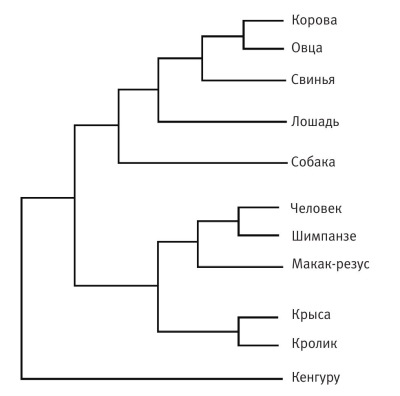
Древо эволюционных изменений по Д. Пенни
Объем генетических последовательностей, накопленный за последнее время, вообще устраняет всякие сомнения. Результаты сравнения генетических деревьев куда более убедительно, чем данные палеонтологии (сами по себе убедительные), сходятся стремительно и безусловно к единому великому древу жизни. На иллюстрации изображено дерево млекопитающих, которых изучал Пенни и его коллеги, но уже с учетом консенсусного голосования гигантского количества частей их генома. Именно постоянная согласованность результатов исследований разных генов позволяет нам быть уверенными не только в точности построения самого дерева, но и в самом факте эволюции.
Если молекулярная генетика и дальше будет развиваться экспоненциально, то к 2050 году секвенирование генома любого существа будет занимать не больше времени и стоить не дороже, чем сейчас измерение температуры или кровяного давления. Почему я считаю, что генетические технологии развиваются по экспоненте? Можно ли измерить скорость их развития? В кибернетике существует аналогичное наблюдение — закон Мура, названный в честь одного из основателей компании «Интел». Поскольку существуют разные метрики производительности компьютеров, есть несколько взаимосвязанных формулировок этого закона. Одна из них гласит: количество элементов, которые можно разместить на интегральной микросхеме, удваивается в течение 18–24 месяцев. Это эмпирический закон, выведенный на основе накопленных данных. Закон Мура выполняется уже около пятидесяти лет и, по мнению многих экспертов, останется в силе по меньшей мере еще несколько десятилетий. Другие формулировки закона Мура относятся к скорости вычислений, объему памяти и другим характеристикам в расчете на единицу стоимости, и все они носят экспоненциальный характер. Еще Дарвин продемонстрировал, что экспоненциальный рост ведет к удивительным результатам. С помощью своего сына Джорджа, математика, он показал, что слон, довольно медленно размножающееся животное, всего за несколько столетий неограниченного экспоненциального размножения может заполнить своими потомками планету. Конечно, слоны не размножаются экспоненциально: их размножение ограничено соревнованием за пищу и пространство, болезнями и другими факторами. Собственно, именно это — в какой момент естественный отбор начинает действовать — хотел показать Дарвин.
Закон Мура действует по крайней мере последние полвека. Никто не знает почему, но компьютерная мощность увеличивается экспоненциально, а дарвиновские слоны размножаются по экспоненте только в теории. Я предположил, что аналогичный закон должен действовать и в отношении генной инженерии. Я поделился своим предположением с Джонатаном Ходжкином, оксфордским профессором генетики, моим бывшим студентом. К моей радости, он уже успел об этом подумать и даже провел необходимые вычисления, оценив стоимость секвенирования ДНК стандартного размера в 1965-м, 1975-м, 1995-м и 2000 году. Я представил, сколько ДНК можно секвенировать за тысячу фунтов стерлингов. Полученные цифры отложил на графике в логарифмическом масштабе, что обычно наглядно для экспоненциально растущих величин (на логарифмической шкале они выглядят как прямые линии). Как и должно быть, все четыре точки Ходжкина лежали почти на прямой. Я подобрал к ним прямую (методом линейной регрессии) и экстраполировал ее на будущее. Когда книга отправлялась в типографию, я показал это место в рукописи профессору Ходжкину и узнал о последнем известном ему достижении в этом направлении — секвенировании в 2008 году генома утконоса. Это само по себе очень интересно, поскольку утконос занимает стратегическое положение на древе жизни: наш общий предок жил 180 миллионов лет назад, почти в три раза раньше, чем вымерли динозавры. Я добавил утконоса в график (отмечен звездочкой) и порадовался, что он оказался близко к предполагаемому положению.
Наклон прямой, построенной на основании зависимости, которую я назвал законом Ходжкина (без его разрешения), чуть более пологий, чем у закона Мура, а время удвоения — чуть больше двух лет против чуть менее двух лет для закона Мура. Генная инженерия сильно зависит от компьютерных технологий, так что естественно ожидать сходства обоих законов.
Стрелками показаны размеры генома разных существ. Проведя прямую от стрелки до пересечения с прямой Ходжкина, можно узнать, когда этот геном можно будет секвенировать за тысячу нынешних фунтов стерлингов. Генома дрожжей нужно ждать до 2020 года. В случае генома млекопитающего (с этой точностью все млекопитающие одинаково сложны) моя оценка — чуть раньше 2040 года. Это фантастическая перспектива: всеобъемлющая база данных ДНК всех животных и растений всех царств за приемлемые деньги. Детальное сопоставление ДНК закроет все бреши в знаниях об эволюционном родстве видов и даст нам древо жизни всех живых существ[155]. И как мы только будем рисовать его? Никакого листа не хватит.
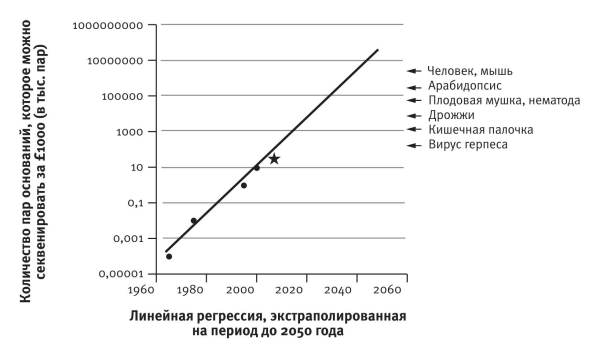
Закон Ходжкина
Самый масштабный проект в этой области был предпринят группой Дэвида Хиллиса (брата разработчика суперкомпьютеров Дэнни Хиллиса). График Хиллиса позволяет представить дерево в более компактном виде посредством наложения на круг. Разрыв, где концы встречаются, почти не виден: он находится между бактериями и архебактериями. Взглянем, например, на сокращенную версию, которую зоолог Клэр Д’Альберто из Мельбурнского университета вытатуировала у себя на спине (интерес Клэр к зоологии поверхностным точно не назовешь). На татуировке (цветная вклейка 25) небольшая выборка — всего 86 видов: именно столько изображено терминальных веточек. Глядя на разрыв на круговом графике, можно представить, что круг развернули.
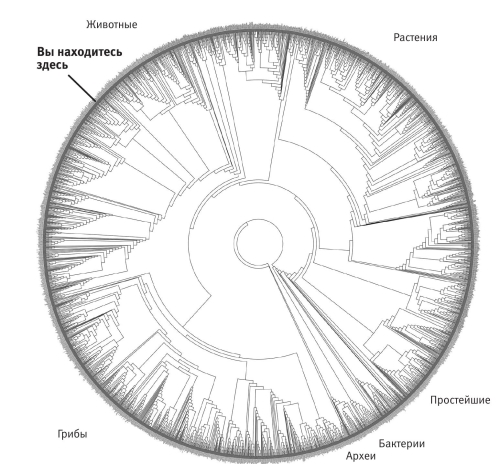
График Хиллиса
Несколько иллюстраций с края — бактерии, простейшие, растения, грибы и четыре других типа животных — выбраны для наглядности. Позвоночные представлены морским драконом — удивительной рыбой, которая защищается, маскируясь под водоросли. График Хиллиса устроен точно так же, однако включает три тысячи видов. Их названия нанесены с внешней стороны круга, но так мелко, что их трудно прочесть, поэтому вид Homo sapiens отмечен особо. Даже столь подробный чертеж покрывает лишь небольшую часть древа жизни: ближайшие родственники человека на нем — крысы и мыши. Число млекопитающих пришлось сильно сократить, чтобы остальные ветви вошли до той же глубины. Попробуйте вообразить себе дерево с десятью миллионами видов вместо трех тысяч у Хиллиса. И, замечу, десять миллионов — не самая вызывающая оценка числа существующих видов. Советую скачать древо Хиллиса на его сайте[156], напечатать и повесить на стену, если, конечно, найдете лист бумаги шириной не менее 137 см.
| <<< Назад Что сделал бы Дарси Томпсон, будь у него компьютер |
Вперед >>> Молекулярные «часы» |
- 11.2. Сопоставление с германской моделью как образцом развитой системы
- Глава I Атомно-молекулярное учение
- 11.2. Сопоставление германской модели ландшафтного планирования с развивающейся российской
- Сопоставление полученного результата с ожидаемым
- Сопоставление с другими раздражителями
- СОПОСТАВЛЕНИЕ ДВУХ СИСТЕМ
- 48. Межмолекулярное взаимодействие.
